Aus Proteopedia
Proteopedia LinkProteopedia Link
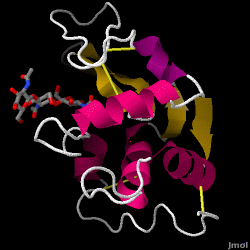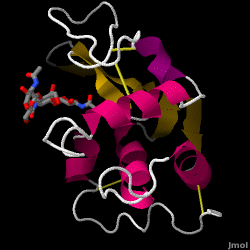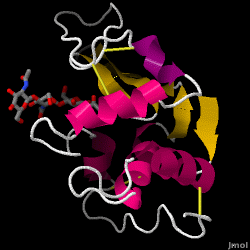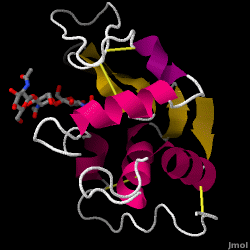
Lysozym – auch bekannt als Muramidase – ist ein leistungsfähiges Enzym, das in Tränen, Speichel und menschlicher Milch reichlich vorhanden ist. Beim Menschen wird es durch das LYZ-Gen kodiert. Da es sich um ein kleines, leicht verfügbares und hochstabiles Protein handelt, wurde seine Funktion und Struktur ausgiebig erforscht.
Einleitung
Lysozym wirkt als unspezifische Abwehr gegen Bakterien und Pilze. Es ist ein Bestandteil des angeborenen Immunsystems und ein wichtiger Bestandteil der Ernährung von Säuglingen zur Abwehr von Durchfallerkrankungen. Es ist ein Enzym, das für seine Fähigkeit bekannt ist, die Polysaccharid-Architektur vieler Arten von Zellwänden abzubauen, normalerweise zum Schutz vor bakteriellen Infektionen. Die Struktur von Hühnereiweiß-Lysozym (HEW), um das es in diesem Artikel geht, ist rechts dargestellt. Die antibakterielle Wirkung von Hühnereiklar wurde erstmals 1909 von Laschtschenko beschrieben. Es wurde von Alexander Fleming, dem Entdecker des Penicillins, charakterisiert und als „Lysozym“ bezeichnet. Die Entdeckung der enzymatischen Aktivität war ein Zufall: Bei einem nicht verwandten Experiment wurde Nasensekret versehentlich in eine Petrischale mit einer Bakterienkultur gegeben, die daraufhin die Ergebnisse einer noch unbekannten enzymatischen Reaktion zeigte. Die Beobachtung dieser unbekannten Reaktion führte zu weiteren Forschungen über die Komponenten dieser Reaktion sowie zur entsprechenden Identifizierung des neu entdeckten „Lysozym“. Im Jahr 1965 bestimmten David C. Phillips und Mitarbeiter die dreidimensionale Struktur von Lysozym mit einer Auflösung von 2 Å. Phillips‘ Arbeit war besonders bahnbrechend, da es ihm gelungen war, die Struktur eines Enzyms mittels Röntgenkristallographie aufzuklären – ein Kunststück, das bis dahin noch nie gelungen war. Phillips‘ Forschungen führten auch zu einer strukturbasierten Hypothese des Wirkmechanismus.
Funktion
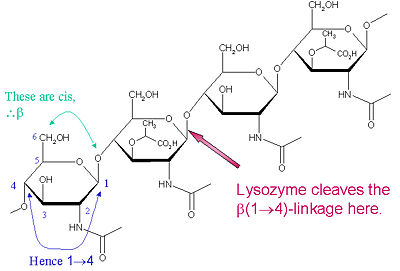
Das bevorzugte Substrat für diesen Spalttyp ist ein (NAG-NAM)₃-Hexasaccharid, innerhalb dessen Substrats die spaltende Zielglykosidbindung, NAM₄-β-O-NAG₅, auftritt. Die einzelnen Hexasaccharid-Bindungseinheiten werden mit A-F bezeichnet, wobei die Präferenz für die Spaltung der glykosidischen Bindung NAM₄-β-O-NAG₅ einer glykosidischen Bindung der Einheit D-E entspricht. cl
Lysozym ist dafür bekannt, dass es bakterielle Zellwände schädigt, indem es die Hydrolyse von 1,4-Beta-Verknüpfungen zwischen N-Acetylmuraminsäure (NAM) und N-Acetyl-D-Glucosamin (NAG) in Peptidoglykanen und zwischen N-Acetyl-D-Glucosamin-Resten in Chitodextrinen. Auf diese Weise ist Lysozym in der Lage, die Zellwände sowohl von Bakterien als auch von Pilzen zu spalten. Der Ort der Spaltung für Lysozym in diesem architektonischen Thema ist die β(1-4)-glykosidische Bindung, die den C1-Kohlenstoff von NAM mit dem C4-Kohlenstoff von NAG verbindet.
Das bevorzugte Substrat für diesen Spalttyp ist ein (NAG-NAM)₃-Hexasaccharid, innerhalb dessen Substrats die spaltende Zielglykosidbindung NAM₄-β-O-NAG₅ auftritt. Die einzelnen Hexasaccharid-Bindungseinheiten werden mit A-F bezeichnet, wobei die Präferenz für die Spaltung der glykosidischen Bindung NAM₄-β-O-NAG₅ einer Präferenz für die Spaltung der glykosidischen Bindung der Einheit D-E entspricht. Je nach Organismus, aus dem Lysozym gewonnen wird, erfolgt die Hydrolyse der Glykosidbindung unter Beibehaltung der Konfiguration am anomeren Kohlenstoff (Hühnereiweiß) oder mit Inversion (Gans, Phage T4).
Lysozym wirkt effizient auf lange (NAG-NAM) oder (NAG) Polymere. Wenn die Kettenlänge kleiner als sechs Monomere wird, sinken die katalytischen Raten erheblich; tatsächlich wirken Trisaccharide als kompetitive Inhibitoren, indem sie an das aktive Zentrum in einem nicht-produktiven Register binden.
Eiweiß-Lysozym (PDB-Code 1hew)
Mechanismus
Die Hydrolyse der glykosidischen Bindungen durch Hühnereiweiß-Lysozym verläuft unter Beibehaltung der Konfiguration. 1953 schlug Koshland vor, dass die Beibehaltung der Konfiguration im Allgemeinen einen Doppelverschiebungsmechanismus impliziert (während die Umkehrung der Konfiguration eine einfache Verschiebung impliziert). Jahrzehntelang wurden zwei konkurrierende mechanistische Hypothesen (Phillips: dissoziativer Mechanismus mit Oxocarbenium-Zwischenstufe; Koshland: zweistufiger assoziativer Mechanismus mit kovalentem Enzymkomplex als Zwischenstufe) in Erwägung gezogen, wobei die Daten aus dem Jahr 2001 eher für die Existenz einer kovalenten Zwischenstufe sprechen. Das Fehlen einer Substratkomplexstruktur hat sicherlich zu Schwierigkeiten bei der Unterscheidung zwischen möglichen Mechanismen beigetragen, ebenso wie die Existenz von zwei verschiedenen Mechanismen (Retention und Konfigurationsumkehr) innerhalb derselben strukturellen Enzymfamilie (z. B. Huhn vs. Gänseenzym).
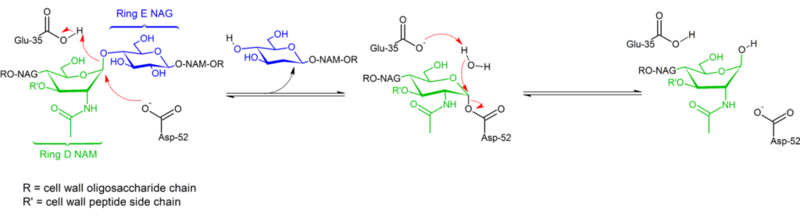
Lysozym hydrolysiert ein Glykosid (daher die familiäre Einordnung von Lysozym als Glykosylase), was der Umwandlung eines Acetals in ein Halbacetal entspricht. Die Reaktion läuft in zwei Schritten ab, wie in der Abbildung oben dargestellt. Im ersten Schritt wirkt Asp 52 als Nukleophil und ein Teil des Zuckers ist die Abgangsgruppe. Im zweiten Schritt fungiert Wasser als Nukleophil und Asp 52 als Abgangsgruppe. In beiden Schritten wird die Konfiguration am anomeren Kohlenstoff invertiert, so dass die Konfiguration insgesamt erhalten bleibt. Glu 35 wirkt im ersten Schritt als Säure (Protonierung des Zuckers an der glykosidischen Bindung, um ihn zu einem besseren Elektrophil zu machen) und im zweiten Schritt als Base (Deprotonierung von Wasser, um ihn zu einem besseren Nukleophil zu machen). Während die Abbildung einige der Zucker in einer Boot-Konformation zeigt, um die Umkehrung der Konfiguration zu betonen, werden diese experimentell nicht beobachtet, sondern eher in einer Stuhl-Konformation gefunden.
Anwendungen von Lysozym
Da Lysozym weithin für seine antibakteriellen und antimykotischen Eigenschaften bekannt ist, hat es eine breite Palette von Anwendungen sowohl in der Biochemie als auch in der Pharmazie. In der Molekularbiologie wird Lysozym häufig im alkalischen Lyseverfahren zur Extraktion und Isolierung von Plasmid-DNA verwendet. Im pharmazeutischen Bereich wird es häufig zur Abtötung grampositiver Bakterien eingesetzt und kann zur Unterstützung der bereits vorhandenen Immunabwehr bei der Bekämpfung bakterieller Infektionen verwendet werden. Dieses Enzym ist besonders wichtig für die Vorbeugung bakterieller Erkrankungen bei Säuglingen. Aufgrund seiner antibakteriellen Eigenschaften kann Lysozym auch in der Lebensmittelindustrie eingesetzt werden, um den Verderb von Lebensmitteln zu verhindern.
Siehe auch
- Lysozym 3D-Strukturen
- Lysozym
- Erhaltende Glykosidhydrolasen
- Molecular Playground/Lysozym
- Benutzer:Judy Voet/Lysozyme
- Lysozyme (arabisch)
- Lysozyme (hebräisch)
- Ragland SA, Criss AK. Von der Bakterientötung zur Immunmodulation: Recent insights into the functions of lysozyme. PLoS Pathog. 2017 Sep 21;13(9):e1006512. doi: 10.1371/journal.ppat.1006512., eCollection 2017 Sep. PMID:28934357 doi:http://dx.doi.org/10.1371/journal.ppat.1006512
- Laschtschenko,P. (1909)Über die keimtötende und entwicklungshemmende Wirkung von Hühnereiweiss. Z. Hyg. Infektionskrankh.,64,419-427.
- Fleming, A. (1922) On a remarkable bacteriolytic element found in tissues and secretions. Proc.Roy.Soc.(London),93,306-317.
- Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Structure of hen egg-white lysozyme. Eine dreidimensionale Fourier-Synthese mit 2 Angström Auflösung. Nature. 1965 May 22;206(986):757-61. PMID:5891407
- Bugg, T. 1997. An Introduction to Enzyme and Coenzyme Chemistry. Blackwell Science Ltd, Oxford
- Earlyliest Solutions for Macromolecular Crystal Structures.
- 7.0 7.1 Blake CC, Johnson LN, Mair GA, North AC, Phillips DC, Sarma VR. Kristallographische Studien über die Aktivität von Hühnereiweiß-Lysozym. Proc R Soc Lond B Biol Sci. 1967 Apr 18;167(1009):378-88. doi:, 10.1098/rspb.1967.0035. PMID:4382801 doi:http://dx.doi.org/10.1098/rspb.1967.0035
- Bild aus: http://www.vuw.ac.nz/staff/paul_teesdale-spittle/essentials/chapter-6/proteins/lysozyme.htm
- Richardson JS. Early ribbon drawings of proteins. Nat Struct Biol. 2000 Aug;7(8):624-5. doi: 10.1038/77912. PMID:10932243 doi:http://dx.doi.org/10.1038/77912
- http://mcdb-webarchive.mcdb.ucsb.edu/sears/biochemistry/tw-enz/lysozyme/HEWL/lysozyme-overview.htm
- Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Structure of hen egg-white lysozyme. Eine dreidimensionale Fourier-Synthese mit 2 Angström Auflösung. Nature. 1965 May 22;206(986):757-61. PMID:5891407
- Johnson LN, Phillips DC. Structure of some crystalline lysozyme-inhibitor complexes determined by X-ray analysis at 6 Angstrom resolution. Nature. 1965 May 22;206(986):761-3. PMID:5840126
- Phillips (1966) Scientific American 215, 76-90
- 14.0 14.1 Vocadlo DJ, Davies GJ, Laine R, Withers SG. Die Katalyse durch Hühnereiweiß-Lysozym erfolgt über ein kovalentes Zwischenprodukt. Nature. 2001 Aug 23;412(6849):835-8. PMID:11518970 doi:10.1038/35090602
- Davies, Withers und Vocadlo (2009) The Chitopentaose Complex of a Mutant Hen Egg-White Lysozyme Displays No Distortion of the -1 Sugar Away from a 4C1 Chair Conformation, Australian Journal of Chemistry 62(6) 528-532
- Koshland, D. E. (1953). Biol. Rev. 28, 416-436
- https://bio.libretexts.org/Bookshelves/Biochemistry/Book%3A_Biochemistry_Online_(Jakubowski)/07%3A_CATALYSIS/B._Mechanisms_of_Enzyme-Catalyzed_Reactions/B2.__Lysozyme
Proteopedia Page Contributors and Editors (was ist das?)
Karsten Theis, Anne Goodling, Michal Harel, Alexander Berchansky, Eric Martz, Karl Oberholser, Joel L. Sussman, Jaime Prilusky, John Ripollone, Daniel Kreider, Judy Voet, John S. de Banzie
Schreibe einen Kommentar