Dalla Proteopedia
proteopedia linkproteopedia link
Il lisozima – noto anche come muramidasi – è un potente enzima che si trova in abbondanza nelle lacrime, nella saliva e nel latte umano. Negli esseri umani, è codificato nel gene LYZ. Poiché si tratta di una proteina piccola, facilmente disponibile e altamente stabile, è stata oggetto di ampie ricerche sulla sua funzione e struttura.
Introduzione
Il lisozima agisce come difesa non specifica contro batteri e funghi. È un componente del sistema immunitario innato ed è una parte importante della dieta di un bambino per evitare la diarrea. È un enzima noto per la sua capacità di degradare l’architettura polisaccaridica di molti tipi di pareti cellulari, normalmente a scopo di protezione contro le infezioni batteriche. La struttura del lisozima dell’albume d’uovo di gallina (HEW), oggetto di questo articolo, è mostrata sulla destra. L’attività antibatterica dell’albume d’uovo di gallina fu descritta per la prima volta da Laschtschenko nel 1909. Fu caratterizzato e chiamato “lisozima” da Alexander Fleming, la stessa persona accreditata per la scoperta della penicillina. La scoperta dell’attività enzimatica avvenne per caso; durante un esperimento non correlato, gocciolamenti nasali furono inavvertitamente introdotti in una capsula di Petri contenente una coltura batterica, la quale coltura esibì di conseguenza i risultati di una reazione enzimatica ancora sconosciuta. L’osservazione di questa reazione sconosciuta portò ad ulteriori ricerche sui componenti di questa reazione e alla corrispondente identificazione del ritrovato “lisozima”. Nel 1965, David C. Phillips e collaboratori determinarono la struttura tridimensionale del lisozima con una risoluzione di 2 Å. Il lavoro di Phillips fu particolarmente innovativo poiché Phillips era riuscito a delucidare con successo la struttura di un enzima tramite la cristallografia a raggi X – un’impresa che non era mai stata compiuta prima. La ricerca di Phillips ha anche portato a un’ipotesi basata sulla struttura del suo meccanismo d’azione.
Funzione
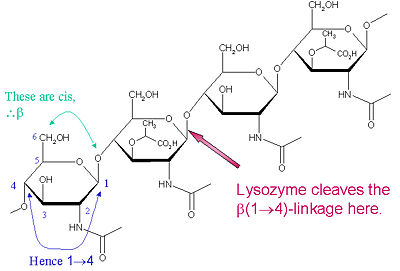
Il particolare substrato di preferenza per questo tipo di clivaggio è un esasaccaride (NAG-NAM)₃, all’interno del quale il substrato si verifica il legame glicosidico target, NAM₄-β-O-NAG₅. Le singole unità di legame esasaccaride sono designate A-F, con NAM₄-β-O-NAG₅ preferenza scissione legame glicosidico corrispondente a un D-E unità legame glicosidico cl
Il lisozima è noto per danneggiare le pareti cellulari batteriche catalizzando l’idrolisi di 1,4-beta-legami tra i residui di acido N-acetilmuramico (NAM) e N-acetil-D-glucosamina (NAG) nel peptidoglicano e tra i residui di N-acetil-D-glucosamina nelle chitodestrine. In questo modo, il lisozima è efficiente nel lisare le pareti cellulari sia dei batteri che dei funghi. La posizione di clivaggio per il lisozima su questo tema architettonico è il legame glicosidico β(1-4) che collega il carbonio C1 di NAM al carbonio C4 di NAG.
Il particolare substrato di preferenza per questo tipo di scissione è un esasaccaride (NAG-NAM)₃, all’interno del quale si verifica il legame glicosidico target, NAM₄-β-O-NAG₅. Le singole unità di legame esasaccaride sono designate A-F, con NAM₄-β-O-NAG₅ preferenza di scissione del legame glicosidico corrispondente a una preferenza di scissione del legame glicosidico unità D-E. A seconda dell’organismo da cui si ottiene il lisozima, l’idrolisi del legame glicosidico procede con mantenimento della configurazione al carbonio anomerico (albume d’uovo di gallina) o con inversione (oca, fago T4).
Il lisozima agisce efficacemente su polimeri lunghi (NAG-NAM) o (NAG). Quando la lunghezza della catena diventa più piccola di sei monomeri, i tassi catalitici diminuiscono sostanzialmente; infatti, i trisaccaridi agiscono come inibitori competitivi legandosi al sito attivo in un registro non produttivo.
Lisozima dell’albume (codice PDB 1hew)
Meccanismo
L’idrolisi dei legami glicosidici da parte del lisozima dell’albume di gallina procede con conservazione della configurazione. Nel 1953, Koshland ha suggerito che in generale, la conservazione della configurazione implica un meccanismo a doppio spostamento (mentre l’inversione della configurazione implica uno spostamento singolo). Per decenni, sono state considerate due ipotesi meccanicistiche concorrenti (Phillips: meccanismo dissociativo con intermedio di ossocarbenio; Koshland: meccanismo associativo a due fasi con complesso enzimatico covalente come intermedio), con i dati del 2001 che fanno pendere la bilancia verso l’esistenza di un intermedio covalente. L’assenza di una struttura del complesso del substrato ha certamente contribuito alla difficoltà di distinguere tra i possibili meccanismi, così come l’esistenza di due meccanismi distinti (ritenzione e inversione di configurazione) all’interno della stessa famiglia strutturale di enzimi (es. enzima di gallina contro oca).
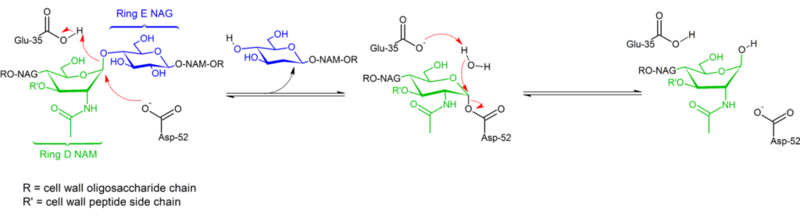
Il lisozima idrolizza un glicoside (da qui la classificazione familiare del lisozima come glicosilasi), che corrisponde alla conversione di un acetale in un emiacetale. La reazione procede in due fasi come mostrato nella figura sopra. Nel primo passo, Asp 52 agisce come nucleofilo e parte dello zucchero è il gruppo di partenza. Nel secondo passo, l’acqua agisce come nucleofilo e Asp 52 agisce come gruppo uscente. Entrambi i passi invertono la configurazione al carbonio anomerico, portando a una conservazione complessiva della configurazione. Glu 35 agisce come un acido nel primo passo (protonando lo zucchero il legame glicosidico per renderlo un migliore elettrofilo) e come una base nel secondo passo (deprotonando l’acqua per renderla un migliore nucleofilo). Mentre la figura mostra alcuni degli zuccheri in una conformazione a barca per sottolineare l’inversione di configurazione, questi non sono osservati sperimentalmente ma si trovano piuttosto in una conformazione a sedia.
Applicazioni del lisozima
Siccome il lisozima è stato ampiamente riconosciuto per le sue proprietà antibatteriche e antifungine, ha un’ampia varietà di usi sia in applicazioni biochimiche che farmaceutiche. In biologia molecolare, il lisozima è spesso usato nella procedura di alcalinizzazione per estrarre e isolare il DNA plasmidico. È ampiamente utilizzato in campo farmaceutico per distruggere i batteri gram-positivi, e può essere usato per sostenere le difese immunitarie già esistenti per combattere le infezioni batteriche. Questo enzima è particolarmente importante per prevenire le malattie batteriche nei bambini. A causa delle sue proprietà antibatteriche, il lisozima può anche essere usato nell’industria alimentare per aiutare a prevenire il deterioramento degli alimenti.
Vedi anche
- Strutture 3D del lisozima
- Lisozima
- Idrolasi glicosidiche di ritenzione
- Molecular Playground/Lysozyme
- Utente:Judy Voet/Lisozima
- Lisozima (arabo)
- Lisozima (ebraico)
- Ragland SA, Criss AK. Dall’uccisione batterica alla modulazione immunitaria: Recenti intuizioni sulle funzioni del lisozima. PLoS Pathog. 2017 Sep 21;13(9):e1006512. doi: 10.1371/journal.ppat.1006512., eCollection 2017 Sep. PMID:28934357 doi:http://dx.doi.org/10.1371/journal.ppat.1006512
- Laschtschenko,P. (1909)Über die keimtötende und entwicklungshemmende Wirkung von Hühnereiweiss. Z. Hyg. Infektionskrankh.,64,419-427.
- Fleming, A. (1922) Su un notevole elemento batteriolitico trovato in tessuti e secrezioni. Proc.Roy.Soc.(London),93,306-317.
- Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Struttura del lisozima di albume di gallina. Una sintesi tridimensionale di Fourier a risoluzione 2 Angstrom. Natura. 1965 maggio 22;206(986):757-61. PMID:5891407
- Bugg, T. 1997. Un’introduzione alla chimica degli enzimi e dei coenzimi. Blackwell Science Ltd., Oxford
- Soluzioni iniziali per le strutture di cristallo macromolecolari.
- 7.0 7.1 Blake CC, Johnson LN, Mair GA, North AC, Phillips DC, Sarma VR. Studi cristallografici dell’attività del lisozima di albume di gallina. Proc R Soc Lond B Biol Sci. 1967 Apr 18;167(1009):378-88. doi:, 10.1098/rspb.1967.0035. PMID:4382801 doi:http://dx.doi.org/10.1098/rspb.1967.0035
- Immagine da: http://www.vuw.ac.nz/staff/paul_teesdale-spittle/essentials/chapter-6/proteins/lysozyme.htm
- Richardson JS. I primi disegni a nastro delle proteine. Nat Struct Biol. 2000 Aug;7(8):624-5. doi: 10.1038/77912. PMID:10932243 doi:http://dx.doi.org/10.1038/77912
- http://mcdb-webarchive.mcdb.ucsb.edu/sears/biochemistry/tw-enz/lysozyme/HEWL/lysozyme-overview.htm
- Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Struttura del lisozima di albume di gallina. Una sintesi tridimensionale di Fourier a risoluzione 2 Angstrom. Natura. 1965 maggio 22;206(986):757-61. PMID:5891407
- Johnson LN, Phillips DC. Struttura di alcuni complessi cristallini lisozima-inibitore determinato da analisi a raggi X a 6 risoluzione Angstrom. Natura. 1965 maggio 22;206(986):761-3. PMID:5840126
- Phillips (1966) Scientific American 215, 76-90
- 14.0 14.1 Vocadlo DJ, Davies GJ, Laine R, Withers SG. La catalisi da parte del lisozima di albume di gallina procede attraverso un intermedio covalente. Natura. 2001 Aug 23;412(6849):835-8. PMID:11518970 doi:10.1038/35090602
- Davies, Withers e Vocadlo (2009) Il complesso Chitopentaose di un mutante Hen Egg-White Lysozyme Displays No Distortion of the -1 Sugar Away from a 4C1 Chair Conformation, Australian Journal of Chemistry 62(6) 528-532
- Koshland, D. E. (1953). Biol. Rev. 28, 416-436
- https://bio.libretexts.org/Bookshelves/Biochemistry/Book%3A_Biochemistry_Online_(Jakubowski)/07%3A_CATALYSIS/B._Mechanisms_of_Enzyme-Catalyzed_Reactions/B2.__Lysozyme
Proteopedia Page Contributors and Editors (what is this?)
Karsten Theis, Anne Goodling, Michal Harel, Alexander Berchansky, Eric Martz, Karl Oberholser, Joel L. Sussman, Jaime Prilusky, John Ripollone, Daniel Kreider, Judy Voet, John S. de Banzie
Lascia un commento