From Proteopedia
proteopedia linkproteopedia link
 >
>
Lysozyme – também conhecida como muramidase – é uma enzima poderosa encontrada em abundância em lágrimas, saliva e leite humano. Em humanos, ela é codificada no gene LYZ. Por ser uma proteína pequena, facilmente disponível e altamente estável, ela tem sido objeto de extensa pesquisa quanto à sua função e estrutura.
Introdução
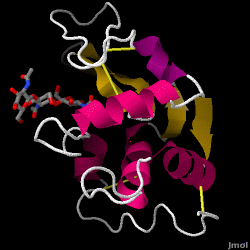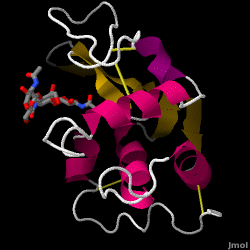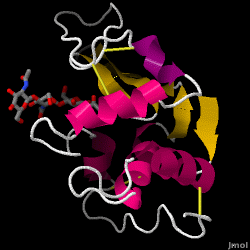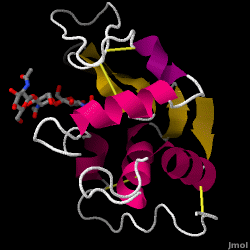
Lysozyme atua como uma defesa não específica contra bactérias e fungos. É um componente do sistema imunológico inato, e é uma parte importante da dieta de uma criança para evitar diarréia. É uma enzima conhecida pela sua capacidade de degradar a arquitectura polissacarídica de muitos tipos de paredes celulares, normalmente com o objectivo de protecção contra infecções bacterianas. A estrutura da lisozima da clara de ovo (HEW), o foco deste artigo, é mostrado à direita. A atividade antibacteriana da clara de galinha foi descrita pela primeira vez por Laschtschenko em 1909. Foi caracterizada e denominada “lisozima” por Alexander Fleming, a mesma pessoa creditada pela descoberta da penicilina. A descoberta da actividade enzimática foi por acidente; durante a experiência não relacionada, foram introduzidos, inadvertidamente, gotejamentos nasais numa placa de petri contendo uma cultura bacteriana, cuja cultura exibia, consequentemente, os resultados de uma reacção enzimática ainda desconhecida. A observação desta reação desconhecida levou a pesquisas adicionais sobre os componentes desta reação, bem como à correspondente identificação da recém-descoberta “lisozima”. Em 1965, David C. Phillips e colegas de trabalho determinaram a estrutura tridimensional da lisozima com uma resolução de 2 Å . O trabalho de Phillips foi especialmente inovador, uma vez que Phillips tinha conseguido elucidar com sucesso a estrutura de uma enzima via cristalografia de raios X – um feito que nunca antes tinha sido alcançado. A pesquisa de Phillips também levou a uma hipótese baseada na estrutura de seu mecanismo de ação.
Função
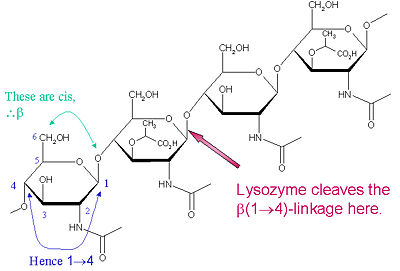
O substrato particular de preferência para este tipo de clivagem é um (NAG-NAM)₃ hexasacarídeo, dentro do qual o substrato ocorre a ligação glicosídica alvo de clivagem, NAM₄-β-O-NAG₅. As unidades de ligação hexassacarídeo individuais são designadas por A-F, com NAM₄-β-O-NAG₅ preferência pela clivagem da ligação glicosídica correspondente a uma unidade D-E de ligação glicosídica cl
Lysozyme é conhecida por danificar as paredes celulares bacterianas ao catalisar a hidrólise de 1,4-beta-ligações entre o ácido N-acetilmurâmico (NAM) e os resíduos de N-acetil-D-glucosamina (NAG) no peptidoglicano, e entre os resíduos de N-acetil-D-glucosamina na quitodextrina. Desta forma, a lisozima é eficiente na lisagem das paredes celulares tanto das bactérias como dos fungos. A localização da clivagem para lisozima neste tema arquitetônico é o β(1-4) ligação glicosídica ligando o carbono C1 do NAM ao carbono C4 do NAG.
O substrato particular de preferência para este tipo de clivagem é um (NAG-NAM)₃ hexasacarídeo, dentro do qual ocorre o substrato da ligação glicosídica alvo da clivagem, NAM₄-β-O-NAG₅. As unidades de ligação hexassacarídeo individuais são designadas A-F, com NAM₄-β-O-NAG₅ preferência pela clivagem da ligação glicosídica correspondente a uma unidade D-E preferência pela clivagem da ligação glicosídica. Dependendo do organismo do qual a lisozima é obtida, a hidrólise da ligação glicosídica procede com retenção da configuração no carbono anomérico (clara de galinha) ou com inversão (ganso, fago T4).
Lisozima atua eficientemente em polímeros longos (NAG-NAM) ou (NAG). Como o comprimento da cadeia fica menor que seis monômeros, as taxas catalíticas caem substancialmente; na verdade, os trissacarídeos atuam como inibidores competitivos ligando-se ao local ativo em um registro não-produtivo.
Lisozima de clara de ovo (código PDB 1hew)
Mecanismo
Hidrólise de ligações glicosídicas por lisozima de clara de ovo procede com a retenção da configuração. Em 1953, Koshland sugeriu que, em geral, a retenção da configuração implica um mecanismo de duplo deslocamento (enquanto que a inversão da configuração implica um único deslocamento). Durante décadas, duas hipóteses mecanicistas concorrentes (Phillips: mecanismo dissociativo com intermediário oxocarbênio; Koshland: mecanismo associativo de dois passos com complexo enzimático covalente como intermediário) foram consideradas, com dados de 2001 inclinando a escala para a existência de um intermediário covalente. A ausência de uma estrutura complexa de substrato certamente contribuiu para dificuldades na distinção entre possíveis mecanismos, assim como a existência de dois mecanismos distintos (retenção e inversão de configuração) dentro da mesma família estrutural de enzimas (por exemplo hen vs goose enzyme).
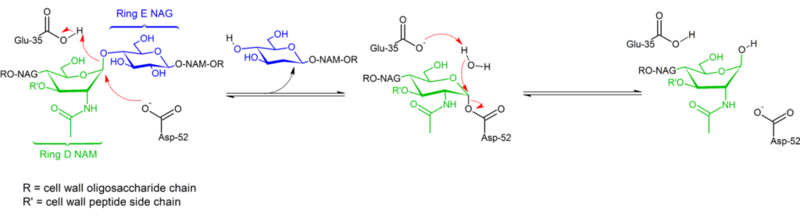
Lisozima hidrolisa um glicosídeo (daí a classificação familiar da lisozima como uma glicosilase), que corresponde à conversão de um acetal em um hemiacetal. A reação prossegue em dois passos, como mostra a figura acima. No primeiro passo, a Asp 52 age como nucleófilo e parte do açúcar é o grupo de saída. No segundo passo, a água atua como nucleófilo e a Asp 52 atua como grupo de saída. Os dois passos invertem a configuração no carbono anomérico, levando a uma retenção geral da configuração. O Glu 35 atua como um ácido no primeiro passo (protonando o açúcar a ligação glicosídica para torná-lo um melhor eletrofilo) e como base no segundo passo (desprotonificando a água para torná-lo um melhor nucleófilo). Enquanto a figura mostra alguns dos açúcares em uma conformação de barco para enfatizar a inversão de configuração, estes não são observados experimentalmente, mas são encontrados em uma conformação de cadeira.
Aplicações da lisozima
Desde que a lisozima tem sido amplamente reconhecida por suas propriedades antibacterianas e antifúngicas, ela tem uma grande variedade de usos tanto em aplicações bioquímicas como farmacêuticas. Na biologia molecular, a lisozima é frequentemente utilizada no procedimento de extracção e isolamento do ADN plasmídeo. É amplamente utilizada no campo farmacêutico para destruir bactérias gram-positivas, e pode ser usada para apoiar defesas imunológicas já existentes para combater infecções bacterianas. Esta enzima é particularmente importante para a prevenção de doenças bacterianas em bebés. Devido às suas propriedades antibacterianas, a lisozima também pode ser utilizada na indústria alimentar para ajudar a prevenir a deterioração dos alimentos.
Ver também
- Estruturas de lisozima 3D
- Lisozima
- Hidrolases Glicosídicas de Retenção
- Playground Molecular/Lisozima
- Utilizador:Judy Voet/Lisozima
- Lisozima (árabe)
- Lisozima (hebraico)
- Ragland SA, Criss AK. Da matança bacteriana à modulação imunológica: Conhecimentos recentes sobre as funções da lisozima. PLoS Pathog. 2017 Set 21;13(9):e1006512. doi: 10.1371/journal.ppat.1006512., eCollection 2017 Sep. PMID:28934357 doi:http://dx.doi.org/10.1371/journal.ppat.1006512
- Laschtschenko,P. (1909)Über die keimtötende und entwicklungshemmende Wirkung von Hühnereiweiss. Z. Hyg. Infektionskrankh.,64,419-427.
- Fleming, A. (1922) Sobre um notável elemento bacteriolítico encontrado nos tecidos e secreções. Proc.Roy.Soc.(London),93,306-317,
- Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Estrutura da lisozima de clara de ovo de galinha. Uma síntese tridimensional de Fourier com resolução de 2 Angstrom. Natureza. 1965 Maio 22;206(986):757-61. PMID:5891407
- Bugg, T. 1997. An Introduction to Enzyme and Coenzyme Chemistry. Blackwell Science Ltd., Oxford
- Primeiras Soluções para Estruturas de Cristal Macromoleculares.
- 7.0 7.1 Blake CC, Johnson LN, Mair GA, North AC, Phillips DC, Sarma VR. Estudos cristalográficos da atividade da lisozima de clara de ovo de galinha. Proc R Soc Lond B Biol Sci. 1967 Abr 18;167(1009):378-88. doi:, 10.1098/rspb.1967.0035. PMID:4382801 doi:http://dx.doi.org/10.1098/rspb.1967.0035
- Imagem de: http://www.vuw.ac.nz/staff/paul_teesdale-spittle/essentials/chapter-6/proteins/lysozyme.htm
- Richardson JS. Desenhos iniciais de fitas de proteínas. Nat Struct Biol. 2000 Ago;7(8):624-5. doi: 10.1038/77912. PMID:10932243 doi:http://dx.doi.org/10.1038/77912
- http://mcdb-webarchive.mcdb.ucsb.edu/sears/biochemistry/tw-enz/lysozyme/HEWL/lysozyme-overview.htm
- Blake CC, Koenig DF, Mair GA, North AC, Phillips DC, Sarma VR. Estrutura da lisozima de clara de ovo de galinha. Uma síntese tridimensional de Fourier com resolução de 2 Angstrom. Natureza. 1965 Maio 22;206(986):757-61. PMID:5891407
- Johnson LN, Phillips DC. Estrutura de alguns complexos de inibidores de lisozima cristalina determinados pela análise de raios X com resolução de 6 Angstrom. Natureza. 1965 Maio 22;206(986):761-3. PMID:5840126
- Phillips (1966) Scientific American 215, 76-90
- 14.0 14.1 Vocadlo DJ, Davies GJ, Laine R, Withers SG. Catalysis by hen egg-white lysozyme proceeds via a covalent intermediate. Natureza. 2001 Ago 23;412(6849):835-8. PMID:11518970 doi:10.1038/35090602
- Davies, Withers e Vocadlo (2009) The Chitopentaose Complex of a Mutant Hen Egg-White Lysozyme Displays No Distortion of the -1 Sugar Away from a 4C1 Chair Conformation, Australian Journal of Chemistry 62(6) 528-532
- Koshland, D. E. (1953). Biol. Rev. 28, 416-436
- https://bio.libretexts.org/Bookshelves/Biochemistry/Book%3A_Biochemistry_Online_(Jakubowski)/07%3A_CATALYSIS/B._Mecanismos_de_Reacções_de_Enzimas-Catalizadas_B2.__Lysozyme
Contribuintes e Editores da Proteopedia Page (o que é isto?)
Karsten Theis, Anne Goodling, Michal Harel, Alexander Berchansky, Eric Martz, Karl Oberholser, Joel L. Sussman, Jaime Prilusky, John Ripollone, Daniel Kreider, Judy Voet, John S. de Banzie
Deixe uma resposta